Combinación del número de emparejamientos
Lo que busco es un tipo de colección donde cada elemento de la colección es un par de valores. Cada valor del par puede tener su propio tipo (como el ejemplo de String y Integer de arriba), que se define en el momento de la declaración.
En Java 8, se puede utilizar la clase javafx.util.Pair para crear un par inmutable y serializable. Esta clase permite claves y valores nulos. (En Java 9, esta clase está incluida en el módulo javafx.base). EDIT: A partir de Java 11, JavaFX se ha desacoplado del JDK, por lo que necesitarás el artefacto adicional de maven org.openjfx:javafx-base.
A partir de Java 6, se puede utilizar la clase AbstractMap.SimpleImmutableEntry para un par inmutable, o AbstractMap.SimpleEntry para un par cuyo valor puede cambiarse. Estas clases también permiten claves y valores nulos, y son serializables.
o bien, con la siguiente verbosidad (que, a diferencia de la clase listada en la respuesta aceptada, protege contra NullPointerExceptions, y tiene una implementación robusta de hashCode() idéntica a la de Records1):

Cuántas formas de emparejar a 2n estudiantes
“Emparejamiento” es un término utilizado para describir a dos o más personas juntas. El “shipping” es el acto de apoyar un emparejamiento o una relación entre dos personajes, pero el término “emparejamiento” tiende a utilizarse para definir cualquier grupo de dos personas juntas en un sentido genérico irrelevante para el “shipping”; por ejemplo, algunos artistas fanáticos describen dos personajes en una obra de arte sin trasfondo romántico/sexual como una representación de emparejamiento. También existe un término llamado One True Pairing (OTP) que se utiliza para describir el emparejamiento favorito de alguien.
Algunos creadores e incluso fans de los Personajes Originales los emparejan con personajes canónicos, incluso si dicha relación no está explícitamente respaldada por el creador del personaje. Estos emparejamientos se reconocen aquí en las páginas de sus respectivos personajes originales, pero no reciben sus propios nombres de portmaneu o páginas en esta wiki.
4 opciones cuántas combinaciones
El emparejamiento de bases no canónico se produce cuando las nucleobases se unen por enlace de hidrógeno, o emparejamiento de bases, entre sí en esquemas distintos a los pares de bases Watson-Crick estándar (que son adenina (A) — timina (T) en el ADN, adenina (A) — uracilo (U) en el ARN, y guanina (G) — citosina (C) tanto en el ADN como en el ARN). Hay tres tipos principales de pares de bases no canónicas: los estabilizados por enlaces de hidrógeno polares, los que tienen interacciones entre grupos C-H y O/N, y los que tienen enlaces de hidrógeno entre las propias bases[1] Los primeros pares de bases no canónicas descubiertos son los pares de bases Hoogsteen, que fueron descritos por primera vez por el bioquímico estadounidense Karst Hoogsteen.
Los emparejamientos de bases no canónicas se producen habitualmente en la estructura secundaria del ARN (por ejemplo, el emparejamiento de G con U) y en el reconocimiento del ARNt. Suelen ser menos estables que los emparejamientos de bases estándar[2] La presencia de pares de bases no canónicos en el ADN de doble cadena da lugar a una doble hélice interrumpida[3].
James Watson y Francis Crick publicaron la estructura de doble hélice del ADN y propusieron los pares de bases canónicos de Watson-Crick en 1953[4]. Diez años más tarde, en 1963, Karst Hoogsteen informó de que había utilizado la difracción de rayos X de un solo cristal para investigar estructuras de pares de bases alternativas, y encontró una estructura alternativa para el par de nucelobases adenina-timina en la que la purina (A) adopta una conformación alternativa con respecto a la pirimidina (T)[5]. [5] Cinco años después de que Hoogsteen propusiera el par de bases A-T Hoogsteen, se informó de los espectros de dispersión óptica rotativa que proporcionaban pruebas de un par de bases G-C Hoogsteen. [6] El par de bases G-C Hoogsteen se observó por primera vez mediante cristalografía de rayos X años más tarde, en 1986, al co-cristalizar el ADN con triostina A (un antibiótico) 7] Finalmente, tras años de estudio de los pares de bases Watson-Crick y Hoogsteen, se ha determinado que ambos se dan de forma natural en el ADN, y que existen en equilibrio el uno con el otro; las condiciones en las que existe el ADN determinan, en última instancia, qué forma se verá favorecida. [8] Desde que se determinaron las estructuras de los pares de bases canónicas Watson-Crick y Hoogsteen no canónicas, se han presentado y descrito muchos otros tipos de pares de bases no canónicas.
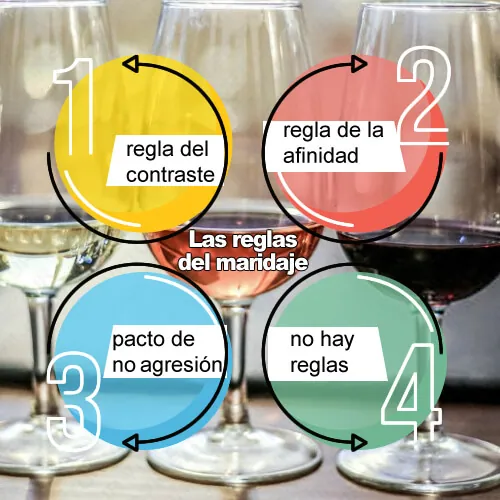
Maridaje de alimentos y vinos
El vino y el queso son compañeros de cama bien conocidos, pero si es usted principiante puede parecerle desalentador decidir exactamente qué vino elegir para cada queso. Esta guía le ayudará rápidamente a empezar a maridar vino y queso como un profesional.
La mayoría de los principiantes en el maridaje de vinos y quesos comenzarán con un queso en particular o buscarán vinos para una tabla de quesos. Así que hemos desglosado las cosas por tipo de queso. (Para conocer los vinos que pueden combinarse con su queso favorito, consulte más abajo)
Los quesos duros son el cheddar, el comté, el parmigiano reggiano y el manchego. Son el tipo de queso más fácil de maridar con vino: un tinto de cuerpo medio, como un cabernet sauvignon o un rioja, será probablemente el maridaje más agradable para la mayoría de la gente, pero pruebe la combinación de cheddar y chardonnay.
Los quesos blandos van desde los quesos para untar como el Philadelphia hasta los quesos de corteza blanca semiblanda como el brie y el camembert. A mí me gustan con un tinto afrutado como un pinot noir o un Beaujolais, pero el rosado también funciona bien con este estilo de queso.
Los quesos azules son el Stilton, el Roquefort y el Gorgonzola. Los maridajes clásicos suelen ser dulces, por ejemplo, Sauternes con Roquefort u oporto con Stilton. Para algo diferente, pruebe con la ginebra de endrinas o el jerez dulce. Y, aunque parezca mentira, incluso la cerveza negra y el queso azul funcionan bien juntos.
